Blog | Temps de lecture 3 minutes
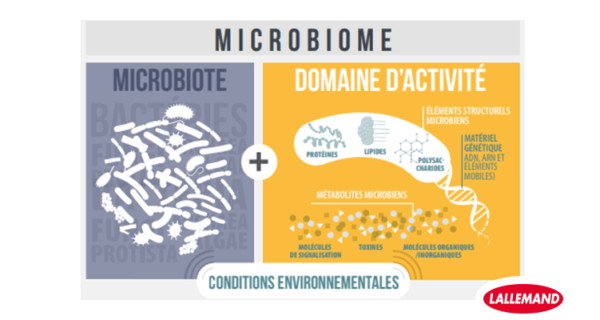
Comment étudier le microbiome ?
Au cours de ces dernières années, les progrès et l’accessibilité accrue aux techniques de séquençage de l’ADN ont permis aux scientifiques de mieux comprendre le microbiome animal. Mais comment cela fonctionne-t-il ?
Qu’est ce que la métagénomique ?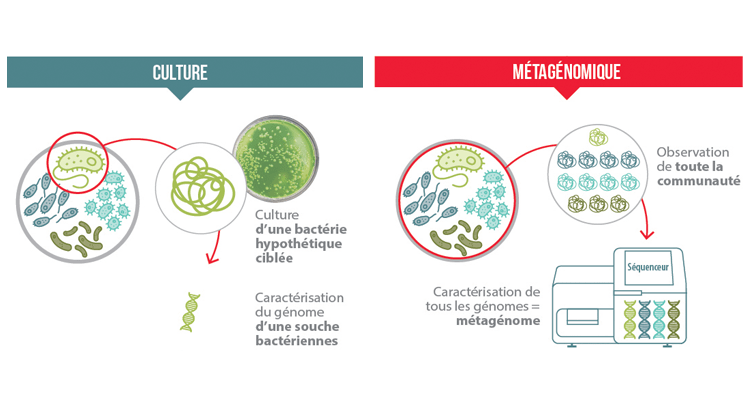
Pour identifier et caractériser les micro-organismes, les microbiologistes utilisaient auparavant des techniques reposant sur la culture microbienne et la microscopie. Ces techniques fournissent une vision limitée d’une communauté microbienne et prennent beaucoup de temps. Récemment, grâce aux progrès accomplis en matière de techniques de séquençage à haut débit, les microbiologistes ont fait un pas de géant avec le développement de la métagénomique.
- La métagénomique s’appuie sur une série de techniques de séquençage et d’outils de bio-informatique pour accéder directement au contenu génétique de communautés entières de micro-organismes (Thomas et al., 2012). La culture et la métagénomique sont complémentaires.
Ce qui caractérise les techniques de métagénomique :
- Elles permettent de détecter des espèces bactériennes rares, ainsi que certaines espèces ne pouvant pas être mises en culture.
- Elles permettent l’analyse simultanée d’un grand nombre d’échantillons microbiens.
- Elles donnent un aperçu de la diversité des populations au sein d’un échantillon.
Ces techniques nécessitent une expertise très spécifique en biostatistique et des moyens bio-informatiques importants pour traduire les millions de séquences d’ADN générées en une composition de population microbienne. Elles s’appuient également sur une base de données spécifique de séquences bactériennes connues, alors qu’une grande partie est encore inconnue à ce jour. Selon le niveau de détail requis et la complexité du schéma expérimental, l’analyse d’un ensemble de données de séquençage peut prendre entre une semaine et plusieurs mois.
De la métagénomique à l’utilisation des codes-barres : l’ARN 16S
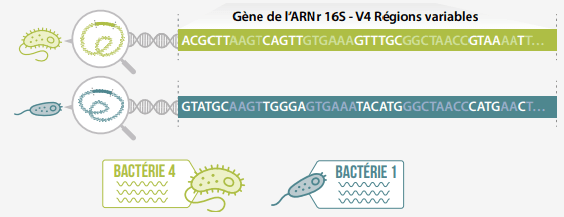
Pour décrire la composition microbienne du microbiote intestinal à l’aide d’une stratégie de séquençage, il n’est pas nécessaire de séquencer le métagénome complet. Une pratique courante consiste à cibler un fragment du génome bactérien, qui est utilisé comme un marqueur ou une sorte de carte d’identité d’une bactérie. Cette approche, appelée séquençage d’amplicons à codes-barres, permet de réduire considérablement le coût et la durée de l’analyse.
- Le gène de la sous-unité de l’ARN ribosomal 16S (gène de l’ARNr 16S) est le gène marqueur le plus utilisé pour décrire la composition des bactéries.
La réduction rapide et importante des coûts du séquençage de nouvelle génération a considérablement accéléré le développement de la métagénomique et la compréhension du microbiote de l’hôte.
Publié Aug 12, 2021 | Mis à jour Dec 4, 2023
Articles liés
Besoin d'informations spécifiques ?
Consulter un expert?